IGV 관련 검색하다가 발견,
2013년 자료인데 재밌는 기능들을 알게 됨.
무려 180여 페이지. PPT 슬라이드니까 체감하기에 그 정돈 아니고,
초보든 어느 정도 아는 사람이든 한번 볼 만하다 싶다.
그 중 눈에 들어온 몇 가지 기능만 우선.
한번에 여러 region 보기. 동시에 몇개까지 볼 수 있는지는 모르겠는데,
targeted seq 결과를 볼 때 유용할 듯.
예전에 genome 전체 말고 몇 곳만 모아서 볼 방법은 없나 하는 생각을 했었는데
이걸로 어느 정도 가능할 것 같다.
여러 gene들을 동시에 볼 수도.
그냥 저 곳에 gene 이름을 늘어놓으면 되는 듯.
멀티뷰 -> 싱글뷰로 돌아가려면.
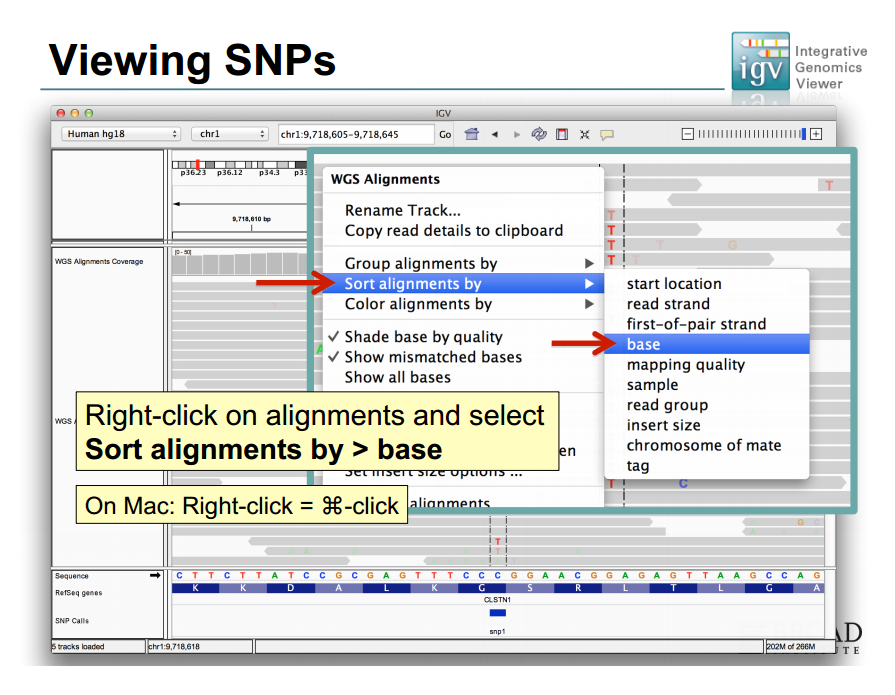
특정 snp에 편리하게 갈 수 있다는데, 'snp1'라는 걸 어찌 추적하지?
'첫 snp'라는 의미도 아닐 거고..
해당 위치를 중앙에 놓고, coverage plot에 빨강, 파랑으로
각 base의 freq를 표시한 건데 아래엔 파란색은 눈에 안 띈다.
정렬을 해보자.
base별로 정렬하고,
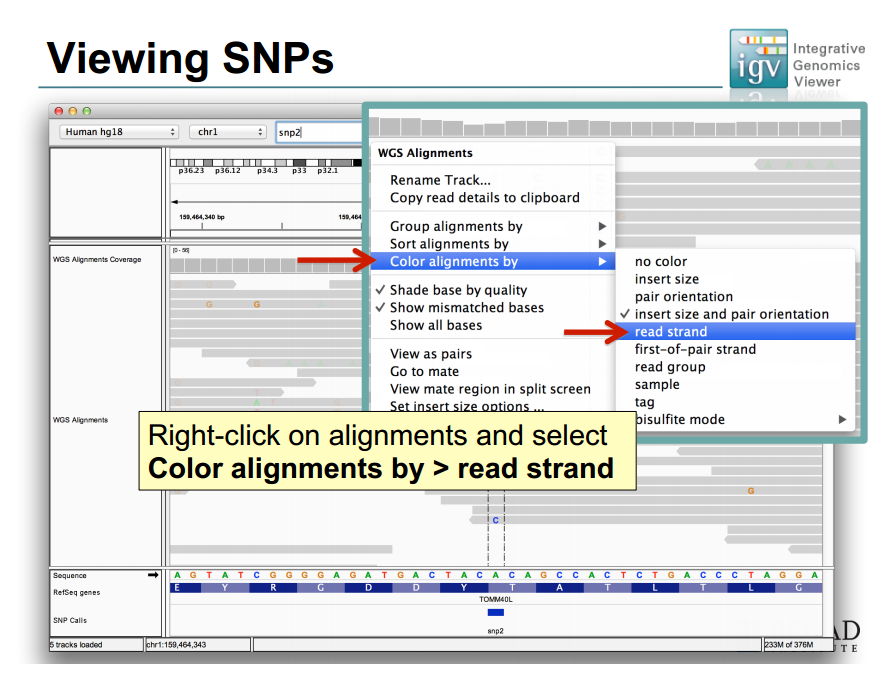
read 매핑상 forward냐 reverse냐에 따라 strand로 정렬
정렬은 했고, 이제 strand를 색깔로 구별하면,
base와 strand를 기준으로 정렬되었고
strand 색깔에 따라 for/rev가 치우져졌는지도 대략 보인다.
그 외에, BAM 파일에 들어있는 read가 너무 많아서 어떤 위치에서 전반적인
매핑 상황 - ref/alt, for/rev 등 -이 잘 안 보이면 downsampling해서 볼 수 있다.
(기본적으로 read 수를 감안해서 그렇게 browsing하게 되어 있는지는 모르겠다)
1. Genome의 inversion된 상황도 확인할 수 있고
2. RNA의 align 상황도 볼 수 있다고 하는데
나중에 다시 봐야겠다.